微软研究院近期宣布了一项突破性进展,推出了一款名为BioEmu-1的人工智能模型,该模型能够预测蛋白质随时间变化的运动和形状演变,为生物医学、药物发现以及结构生物学领域带来了全新的研究视角。
蛋白质,这一生命的基本构建块,在生物体的各种过程中扮演着不可或缺的角色,从肌肉的形成到疾病的防御,无处不在。近年来,科学家利用深度学习技术,在解析蛋白质结构方面取得了显著成就,能够基于氨基酸序列准确预测蛋白质的三维结构。然而,这种预测往往局限于静态结构,就像电影中的一帧画面,无法展现蛋白质的动态特性。
与DeepMind的AlphaFold专注于静态结构预测不同,BioEmu-1致力于模拟蛋白质在不同构象之间的动态转换。这一特性使得BioEmu-1成为理解蛋白质运动机制、设计高效治疗方案的重要工具。虽然AlphaFold 3在结构生物学领域取得了重要进展,改进了蛋白质与其他分子的相互作用模型,但它仍然无法预测蛋白质随时间的动态变化。
BioEmu-1通过生成式深度学习技术,从大型数据集中学习蛋白质结构的模式,并生成与这些模式相符的新样本。它的训练结合了静态蛋白质结构数据、分子动力学模拟数据以及实验稳定性数据,从而能够更全面地理解蛋白质的动态行为。BioEmu-1的核心机制是一个扩散模型,它通过迭代生成蛋白质结构,并根据学习到的约束条件不断提高预测的准确性。
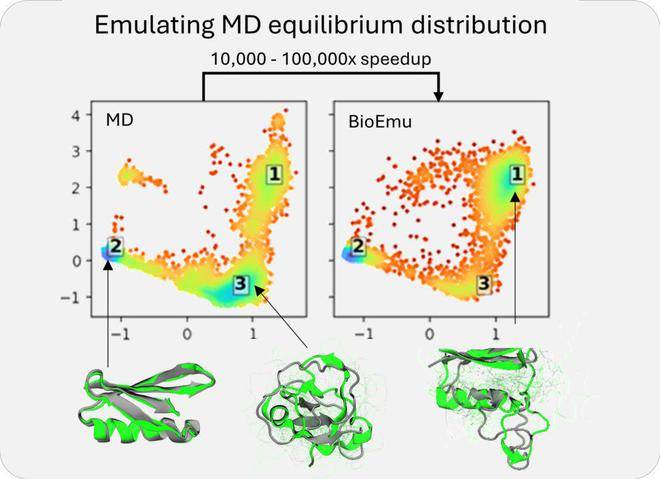
BioEmu-1的输出包括平衡系综的预测和自由能的预测,这些预测对于理解蛋白质的稳定性和功能至关重要。为了训练BioEmu-1,微软研究院使用了三种类型的数据集:AlphaFold数据库的结构数据、广泛的分子动力学模拟数据集以及实验性蛋白质折叠稳定性数据集。通过这些数据集的训练,BioEmu-1能够识别蛋白质序列与多个不同结构之间的映射关系,预测合理的结构变化,并以正确的概率对折叠和未折叠结构进行采样。
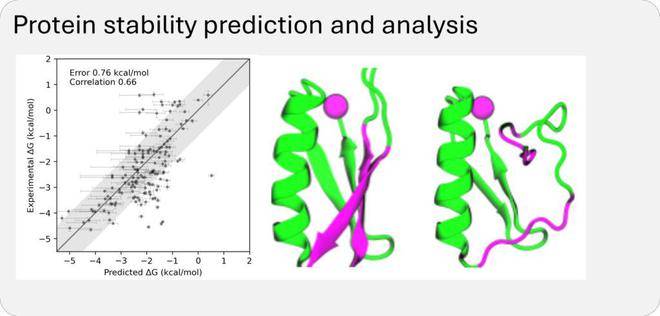
BioEmu-1的高效性也是其一大亮点。它每小时能够生成数千个蛋白质结构样本,这一速度远远超过了传统分子动力学模拟的数周时间。同时,BioEmu-1预测自由能的误差幅度在1 kcal/mol以内,与传统分子动力学模拟相当,但计算成本却显著降低。这意味着研究人员可以更快、更经济地探索蛋白质的动态行为,加速新药的开发进程。
微软研究院的这一创新成果,无疑为蛋白质科学研究开辟了新的道路。BioEmu-1的推出,不仅提高了我们对蛋白质动态行为的理解,还为药物发现和生物医学研究提供了强有力的支持。随着BioEmu-1的广泛应用,我们有理由相信,未来在蛋白质相关领域的研究将取得更加显著的进展。